基于片段的药物发现
基于片段的药物发现方法的主要内容是设计并建立由片段分子组成的化合物库,对化合物库中的分子进行生物活性的筛选从而找到苗头片段分子。再利用X射线晶体学、核磁共振以及质谱技术对这些分子片段与靶蛋白的结合模式与结合强度进行分析,根据这些结构信息对片段分子进行结构优化得到先导化合物。

优点
基于片段的药物发现(FBDD)是确定新先导化合物的重要途径,具有以下优点:
首先,由于片段的分子简单性,它们相对于较大的HTS筛选能更有效地采样。因此,FBDD筛选可以通过包含少至500-1000个选择性良好的筛选片段。
第二,由于片段的体积小,片段相比大分子能更有效地与其靶标结合。已经意识到片段在可开发的药物中有效结合的重要性,可能是由于碎片能够在结合位点采用最佳取向。这种最佳取向对于较大的分子可能更难实现。基于结构导向的优化,FBDD命中的配体可以提供有效的临床候选物。随着越来越多的FBDD衍生分子进入后期临床试验并进入市场,评估FBDD中隐含的结构和特性指导优化,有助于更有效地转化为候选药物。

最后,由于其较小的尺寸,FBDD命中片段的结合亲和力通常比较大命中分子的结合力弱,这种弱亲和力需要使用不同的测定方进行量化。通过使用敏感的生物物理技术,包括NMR、表面等离子体共振、x射线晶体学、热变性、等温滴定量热法等。这些技术能够鉴定亲和力非常弱的配体,且与结合位点的位置无关。因此,生物物理技术有可能识别与变构或隐蔽结合位点相互作用的命中片段。许多生化测定依赖于与特异性结合物的竞争,因此可能无法识别这些独特的结合相互作用。
FBBD方法
1.靶标有效性和片段库的建立;
2.筛选得到可与靶点结合的片段,以及这些片段的结构构象;
3.将命中片断延伸合理的生成、融合和连接成为“候选药物分子”;
4.合成、优化和活性评估。

基于计算的FBDD
为了克服实验性FBDD的局限性,计算方法提供了提高药物发现效率和成功率的替代方法。
计算FBDD过程包括:
(1)构建虚拟片段库;
(2)基于片段的活性位点图和表征;
(3)片段对接进行命中片段的确定;
(4)从命中到先导物再到候选药物的优化。
计算FBDD过程可用于整合的工作流程。该过程中的任何一个步骤也可以用作补充方法辅助实验性FBDD。
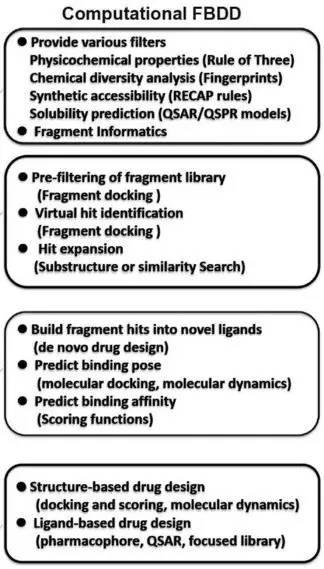
与实验FBDD相比,计算方法有几个优点:
首先,可以通过计算方法构建高质量片段库,设计各种计算过滤器以改善片段库的化学多样性、物理化学性质、溶解度和合成可及性。计算方法也可用于排除含有不想要化学基团的片段并引入药物样片段。
其次,较大的片段数据库可以通过计算工具进行探索。与实验FBDD的低通量性质不同,虚拟碎片筛选可以识别有效的命中,而不使用复杂的检测技术。分子对接可用作预处理工具减少实验FBDD的。
第三,计算方法提供了有效和灵活的优化策略,以改善命中片段的活性和药物相似性。可以使用子结构搜索和相似性搜索来加速命中扩展,并获得二次设计的构效关系(SAR)信息。通过对蛋白质-命中片段复合物的结构信息计算分析,有助于随后优化的最有希望的片段命中的选择。在硅胶SBDD方法中也能够将片段命中积累或组装成具有改善效力和药物相似性的新分子。
研究案例
凝血因子VIIa是一种新的抗血栓形成靶标,与S1口结合的大多数因子VIIa抑制剂包含疏水和阳离子基团,导致不好的膜渗透性和口服吸收。为了发现中性S1靶向抑制剂并改善药代动力学特征,Cheney等研究人员进行了基于片段的虚拟筛选研究。
第一步是构建片段库,可用化学品目录(ACD)数据库基于药物相似、重原子数和可旋转键过滤,产生约18,000个片段;然后通过分子对接程序Glide筛选该片段库,并通过使用蛋白系综对接来考虑蛋白质的柔性。结果,最终选择测定了250种化合物。NMR结合测定鉴定了28个初始命中片段,并且其结合模式通过X射线晶体学测定。

代表性命中片段(Ki = 8.9mM)与凝血因子VIIa S1口袋结合并与氨基酸残基Gly218形成氢键。基于命中片段内酰胺的结合模式,发现了许多具有良好抑制效能和改善渗透性的类似物。如化合物5是细胞可渗透的,并且具有130nM的Ki值。

也许你还有兴趣看:
参考资料:
1.Tian S,Wang J,Li Y,et al.Drug-likeness analysis of traditional Chinese
medicines: prediction of drug-likeness using machine learning approaches[J].Molecular pharmaceutics,2012,9(10):2875-2886.
2.Mendes V,Blundell T L.Targeting tuberculosis using structure-guided fragment-based drug design[J].Drug discovery today,2017,22(3):546-554.
3.DesJarlais R L.6 Using Computational Techniques in Fragment-Based Drug Discovery[J].Methods in enzymology,2011,493:137.
4.Hoffer L,Renaud J P,Horvath D.Fragment-based drug design:computational and experimental state of the art[J].Combinatorial chemistry & high throughput screening,2011,14(6):500-5

