抗
体是免疫系统产生的小蛋白质,可以附着在病毒的特定部位以中和它。
随着科学家们继续与新冠病毒 SARS-CoV-2 作斗争,一种可能的武器是合成抗体;
它与病毒的刺突蛋白结合,从而阻止病毒进入人体细胞。
为了开发成功的合成抗体,研究人员必须准确了解这种附着是如何发生的。具有包含许多折叠的块状 3D 结构的蛋白质可以以数百万种组合形式粘在一起,因此在几乎无数候选物中找到合适的蛋白质复合物非常耗时。
为了简化这一过程,麻省理工学院的研究人员创建了
一个机器学习模型(Equidock),可以直接预测两种蛋白质结合在一起时将形成的复合物。他们的技术比当前最先进的软件方法快 80 到 500 倍,并且经常预测更接近实验观察到的实际结构的蛋白质结构。
该研究以「Independent SE(3)-Equivariant Models for End-to-End Rigid Protein Docking」为题,于 2021 年 9 月 29 日向 ICLR 2022 会议进行盲文投稿。
蛋白质复合物的形成是生物学中的一个核心问题,它参与了细胞的大部分过程,对于药物设计或蛋白质工程等应用至关重要。研究人员处理刚体蛋白质-蛋白质对接,即从单个未结合结构计算预测蛋白质-蛋白质复合物的 3D 结构,假设在结合过程中没有 3D 灵活性。
麻省理工学院的工作人员设计了一种新的成对独立 SE(3)-Equivariant,来预测旋转和平移,以将其中一种蛋白质放置在正确的位置和相对于第二种蛋白质的正确方向。
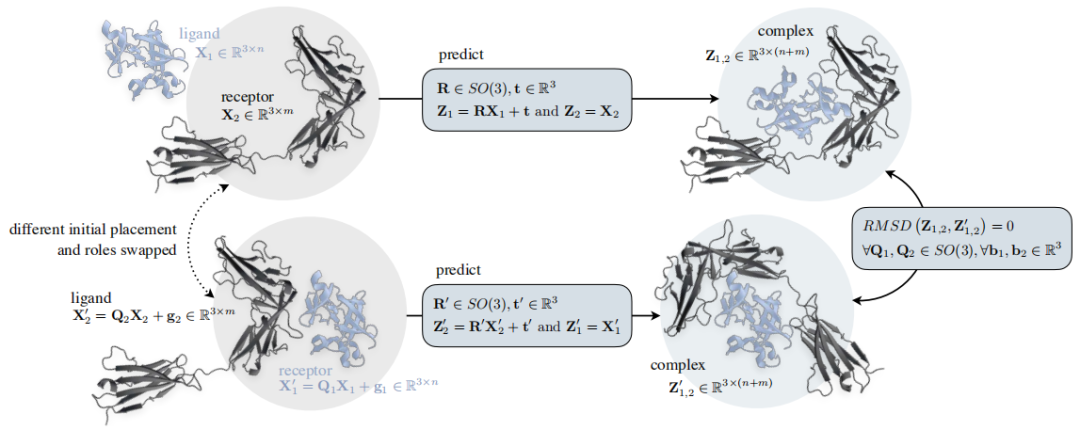
无论两个结构的初始位置如何,该方法在数学上保证预测的复合体总是相同的,避免了昂贵的数据扩充。该模型通过优化传输和可微的 Kabsch 算法使用关键点匹配和对齐来逼近绑定口袋并预测对接姿势。
根据经验,尽管没有使用大量采样、结构细化或模板,但在现有蛋白质对接软件上实现了显著的运行时间改进,并预测了定性合理的蛋白质复杂结构。
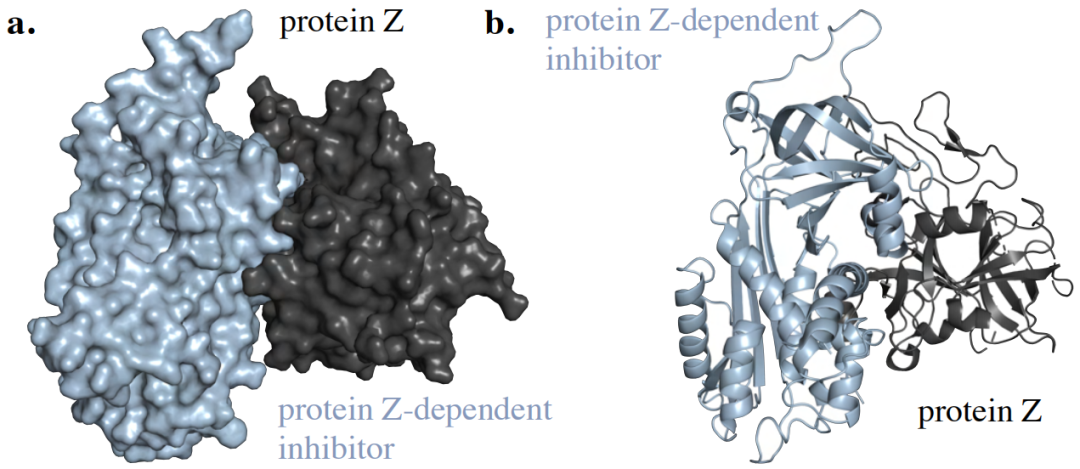
图示:蛋白质复合物 3D 结构的不同视图。(来源:论文)
这项技术可以帮助科学家更好地了解一些涉及蛋白质相互作用的生物过程,如 DNA 复制和修复;它还可以加快开发新药的进程。
「深度学习非常擅长捕捉化学家或生物学家难以通过实验编写的不同蛋白质之间的相互作用。其中一些相互作用非常复杂,人们还没有找到表达它们的好方法。这种深度学习模型可以从数据中学习这些类型的交互。」麻省理工学院计算机科学和人工智能实验室(CSAIL)的博士后 Octavian-Eugen Ganea 说。
图示:与 Equidock 相同的输出保证。(来源:论文)
研究人员开发的名为 Equidock 的模型专注于刚体对接——当两种蛋白质通过在 3D 空间中旋转或平移而附着时发生,但它们的形状不会挤压或弯曲。
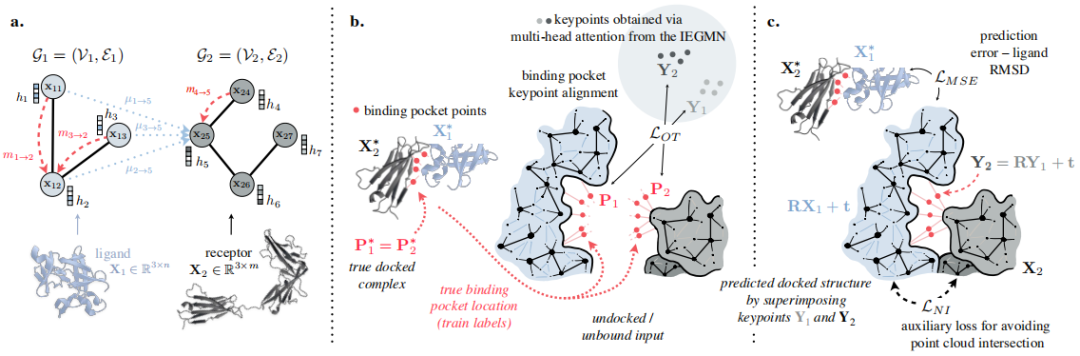
该模型采用两种蛋白质的 3D 结构,并将这些结构转换为可由神经网络处理的 3D 图。蛋白质是由氨基酸链形成的,每个氨基酸都由图中的一个节点表示。
图示:Equidock 架构和损失的详细信息。(来源:论文)
研究人员将几何知识整合到模型中,因此它了解对象在 3D 空间中旋转或平移时会如何变化。该模型还内置了数学知识,可确保蛋白质始终以相同的方式附着,无论它们存在于 3D 空间中的何处。这就是蛋白质在人体中的附着方式。
利用这些信息,机器学习系统识别出两种蛋白质中最有可能相互作用并形成化学反应的原子,称为结合口袋点。然后它使用这些点将两种蛋白质放在一起形成一个复合物。
构建此模型的最大挑战之一是克服缺乏训练数据的问题。Ganea 说,由于存在如此少的蛋白质实验 3D 数据,因此将几何知识纳入 Equidock 尤为重要。
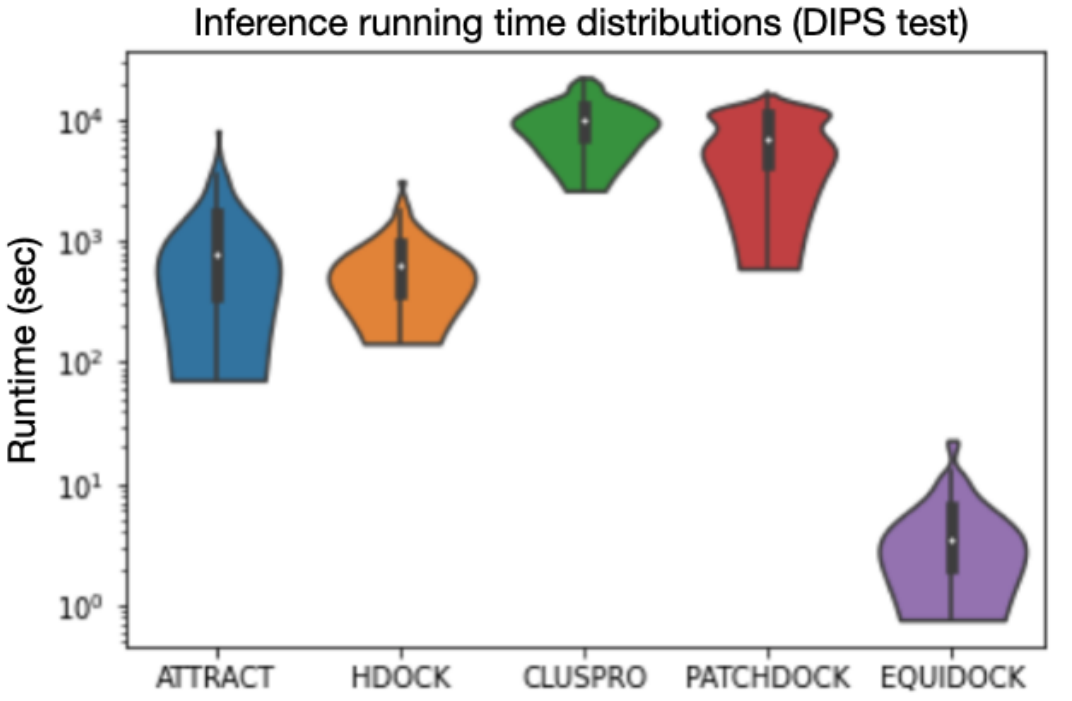
模型训练完成后,研究人员将其与四种软件方法进行了比较。Equidock 能够在一到五秒后预测最终的蛋白质复合物。所有基线都需要更长的时间,从 10 分钟到一个小时或更长时间。
在计算预测蛋白质复合物与实际蛋白质复合物有多接近的质量测量中,Equidock 通常与基线相当,但偶尔表现不佳。
「我们仍然落后于其中一个基线。我们的方法仍然可以改进,并且仍然有用。它可以用于非常大的虚拟筛选,我们想了解数千种蛋白质如何相互作用并形成复合物。我们的方法可用于非常快速地生成一组初始候选者,然后可以使用一些更准确但更慢的传统方法对这些候选者进行微调。」Ganea 说。
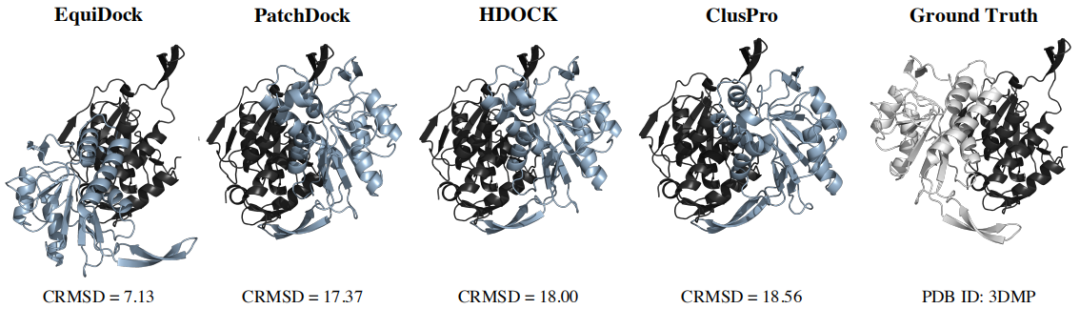
图示:Equidock 成功预测的蛋白质复合物的可视化。(来源:论文)
除了将这种方法与传统模型一起使用之外,该团队还希望将特定的原子相互作用纳入 Equidock 中,以便做出更准确的预测。例如,有时蛋白质中的原子会通过疏水相互作用附着,其中涉及水分子。
未来,他们计划增强 Equidock,以便它可以预测灵活的蛋白质对接。最大的障碍是缺乏训练数据,因此 Ganea 和他的同事正在努力生成可用于改进模型的合成数据。
论文链接:https://openreview.net/forum?id=GQjaI9mLet
相关报道:https://phys.org/news/2022-02-artificial-intelligence-rapidly-proteins.html
人工智能 × [ 生物 神经科学 数学 物理 材料 ]
「ScienceAI」关注人工智能与其他前沿技术及基础科学的交叉研究与融合发展。
欢迎关注标星,并点击右下角点赞和在看。
点击阅读原文,加入专业从业者社区,以获得更多交流合作机会及服务。