作者 | 王永康
审核 | 熊展坤
今天给大家分享清华大学张牧涵团队发表在ICML2022的论文“3DLinker: An E(3) Equivariant Variational Autoencoder for Molecular Linker Design”。本论文专注于一种新型的药物设计问题,即如何生成一种合适的连接器,将两个独立的目标药物分子片段有效地进行结合。为解决该问题,作者提出一种名为3DLinker的条件生成模型。该模型能够基于E(3)等变图变分自编码器预测分子片段结合过程中的锚原子,并生成连接器的原子图以及3D坐标信息。实验结果表明,该模型在分子片段连接器的恢复上有较好效果,同时可以准确预测分子的3D坐标信息。
01背景
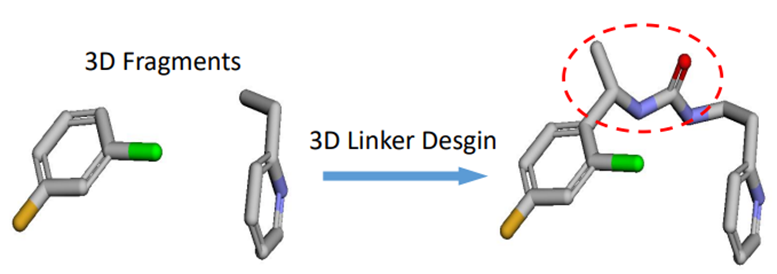
在药物研发中,由于搜索空间的离散性和庞大性,设计包含所需药效团特性的新分子药物仍具有挑战性。近期,一种独特的药物分子将两个具有生物功能的片段分子与接头分子相结合,进而表现出分子接合策略的巨大应用潜力。然而,该策略中的关键点是分子连接器的设计,目前此过程仍依赖于专业的结构生物学家,因此需要一种有效的深度学习方法来解决该问题。 如图1,分子连接器的生成问题可定义为:给定两个具有坐标信息的分子片段图,生成一个包含坐标信息的连接图,用于拼接上述两个片段。同时,生成的连接图坐标需要在空间中与片段相对齐。
图1
02方法
2.1 消息传播 作者设计一种混合特征的消息传播方式(Mixed-Features Message Passing, MF-MP),在每次的消息传播过程中,该方法将不变特征(即分子图的特征嵌入)和等变特征(即分子图的坐标嵌入)进行混合,使其各自的不变性与等变性质得以保存,同时在更新过程中两种特征相互促进。 首先,借助VN-MLP等变神经网络,将不变特征 与等变特征 进行变换,进而混合形成新的中间特征。而后,应用点卷积方法对混合特征进行线性变换。 表示相对位移, 表示相应的核函数,用于将节点的标量距离转换为多维输出向量,从而使消息具有几何感知。最终,对不变特征 采用门控单元,对等变特征 采用等变神经网络来更新节点上的具体消息。
消息获取
消息更新
2.2 编码器
基于上述MF-MP消息传播过程,不变特征 与等变特征 进行充分的信息交互。此处,根据最后一次消息传播的结果,生成两种隐层特征的正态分布描述参数。
2.3 解码器
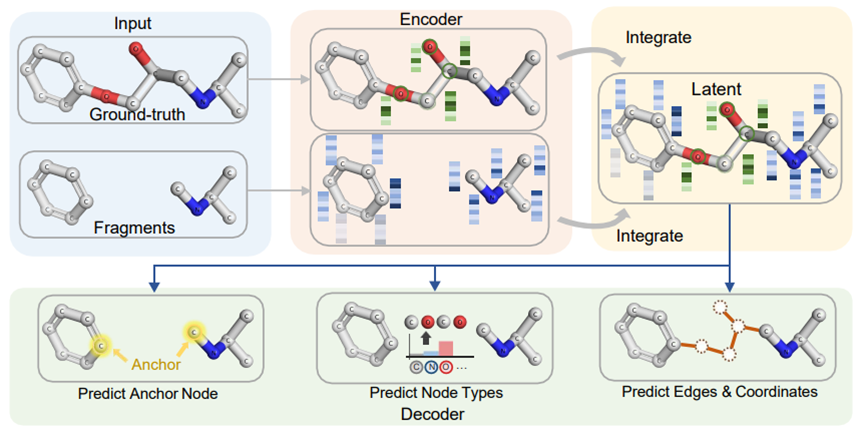
解码器的目的是根据分子片段的不变与等变特征,以顺序方式逐步构造完整的目标连接图过程。具体如下图,首先预测两个分子片段上的锚点信息,以此作为连接器的拼接位置。其次,预测连接器中所有原子的类型。而后,预测连接器中原子之间的边以及相应的3D坐标信息。该过程中以不变与等变特征形式进行连接器的特征描述,进而保证连接器的理论有效性。
图2
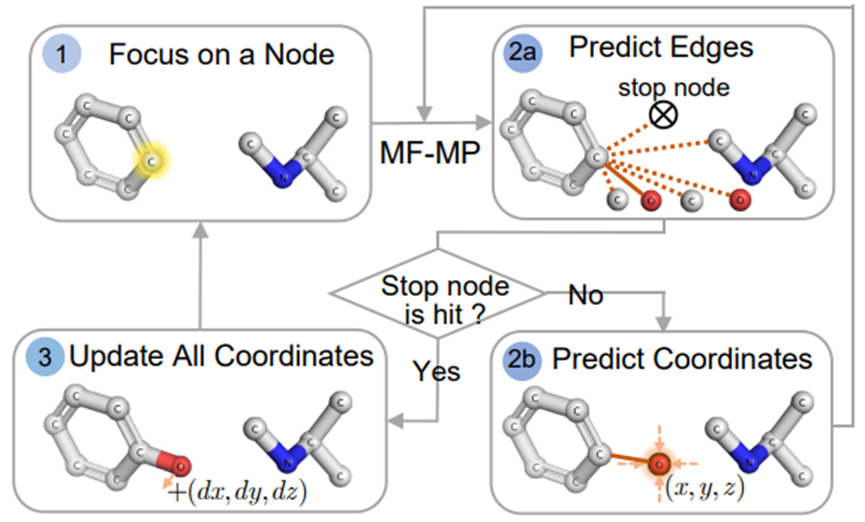
连接器的顺序预测过程如下图:选取目标点,并对其进行邻居的预测(此处以片段的锚点作为预测的起始位点);在目标点与预测点(包括人工定义的终止位点)之间进行边信息采样;若本轮未选中终止节点,则对预测点进行坐标信息的生成,并重复执行预测点过程;若选中终止节点,则更新当前所有节点坐标,并重新选择目标点,执行预测过程,直至连接器中所有节点均被以目标点形式选择过。
图3
03实验结果
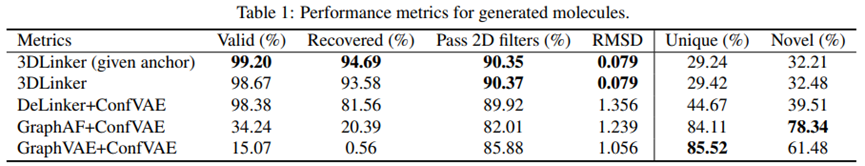
作者选择ZINC数据集作为分子结构来源,从中提取有效的分子片段与连接器信息,并使用RDKit来生成分子的真实三维坐标信息。实验结果表明,3DLinker模型在预测的有效性,恢复率(即描述生成分子与真实分子的恢复比例),2D属性通过率(即合成可及性、环芳香性和泛分析干扰化合物属性),RMSD(即生成分子与真实分子在结构坐标上的差异)指标上,均表现出优秀的性能。
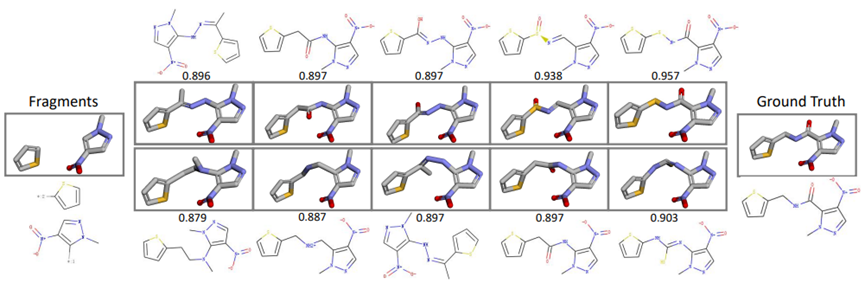
同时,作者随机选取分子片段的连接过程,并可视化与真实分子相似性最高的五种连接器推断,可以发现3DLinker相较于基准DeLinker+ ConfVAE模型,在预测的指标和实际效果上均有较高的优势。
图4
更多实验结果请参考原文。
05总结
针对分子片段的连接问题,作者基于条件变分自编码器,并结合分子的不变特征与等变特征,将分子图的生成与分子三维信息的表征进行整合,从而有效地预测连接器的锚点与边的具体连接状态。该模型在连接器的预测上表现出良好的性能优势,并能够准确获得连接器的有效坐标信息。