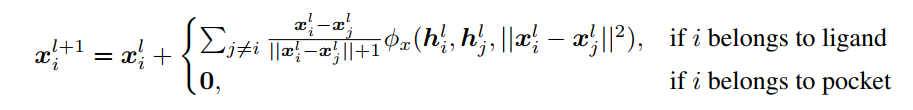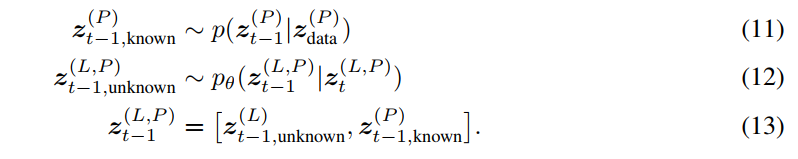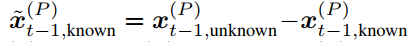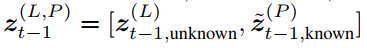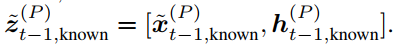编译|陈泽慧
审稿|侯琳琳 今天给大家带来的是洛桑联邦理工学院(EPFL)联合微软研究院Max Welling课题组、牛津、剑桥、康奈尔等团队于2022年10月发表在arxiv上的预印本《Structure-based Drug Design with Equivariant Diffusion Models》。在本文中作者将基于结构的药物设计(SBDD)表述为一个3D条件生成问题,并提出了DiffSBDD,一个 E(3)等变的3D条件扩散模型,它关于平移、旋转、反射和排列等变,并可以根据蛋白质口袋的条件生成新的配体。
简介
合理设计药物分子化合物仍然是生物制药研究中的一个突出挑战。基于结构的药物设计(structure-based drug design, SBDD)旨在生成可以与特定的3D蛋白质结构结合的高亲和力和特异性的小分子配体。然而,SBDD仍然具有很大的挑战性和局限性。传统的SBDD在大规模化学数据库上进行高通量实验或虚拟筛选,但这不仅昂贵且耗时。近些年,生物分子的几何结构建模的快速发展,为基于结构的药物设计提供了一个有希望的方向。尽管利用深度神经网络来替代对接模型已成为常态,但基于深度学习的配体与靶标蛋白结合的设计仍然是一个尚未解决的问题。
在这项工作中,作者为基于结构的药物设计(DiffSBDD)开发了一个等变扩散模型,这是在该方向第一个这样的模型。具体来说,作者将 SBDD 当作3D 条件生成问题,目的是生成对特定蛋白质靶标具有高结合亲和力的多种配体。作者提出了一个 E(3) 等变的3D 条件扩散模型,该模型关于平移、旋转、反射和排列等变。作者介绍了两种策略,即以蛋白质为条件的生成和以蛋白质口袋为条件产生新配体的配体修复生成。具体来说,蛋白质的条件生成是将蛋白质视为一个固定的环境,而配体修复模型则对蛋白质-配体复合物的联合分布进行建模,并在推理过程中修复新配体。同时,作者进一步整理了从binding MOAD导出的实验确定的结合数据集,该数据集补充了常用的合成Crossdocked数据集,以验证提出的模型在现实绑定场景下的性能。实验结果表明,DiffSBDD能够生成新的、多样化的、类药物的配体,并与给定的蛋白质口袋有高结合亲和力。
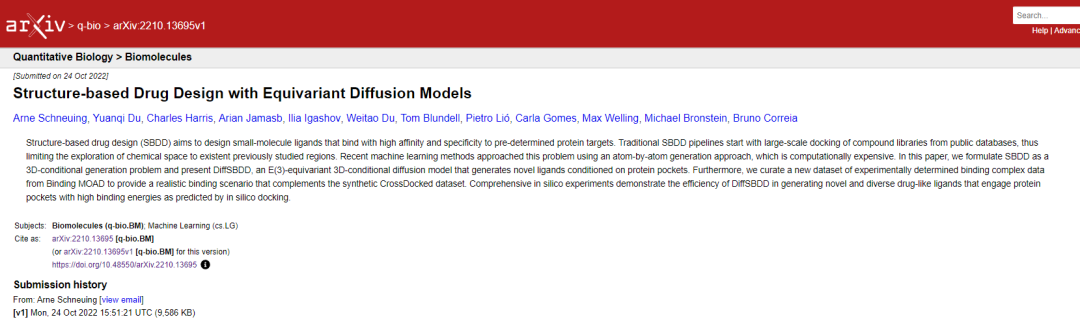
图1:蛋白质条件下的 DiffSBDD。首先模拟前向扩散过程q,以获得在T个时间段内渐进式噪声样本的轨迹。然后训练一个模型p_来反转或去噪这个以目标结构为条件的过程。一旦训练完成,就能够从高斯分布中抽取新的候选药物。原子特征和坐标都在整个过程中扩散。配体 在扩散过程中表示为全连接图(为清楚起见,未显示边缘),并且在生成结束时将共价键添加到生成的点云中。蛋白质以图形式表示,但为了清楚起见,此处显示为表面。
方法
作者利用一个等变DDPM,结合特定的蛋白质靶标来生成分子并结合构象,并将蛋白质和配体点云表示为由 EGNNs 进一步处理的全连接图。作者考虑了两种不同的 3D口袋调节方法:(1)一个条件 DDPM,它在每个去噪步骤中接收一个固定的口袋表示作为背景,以及(2)一个近似配体-口袋对的联合分布的模型,并在推理时与修复相结合。
pocket-conditioned small molecule generation
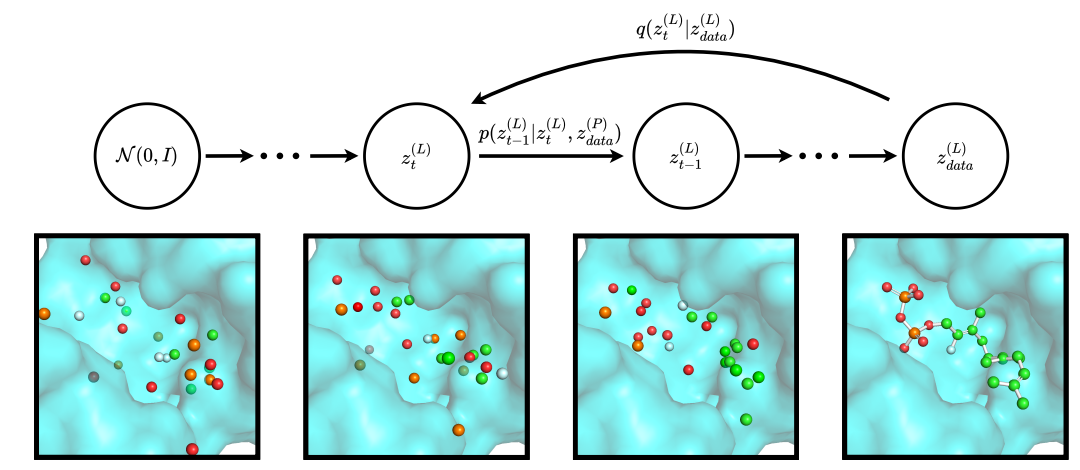
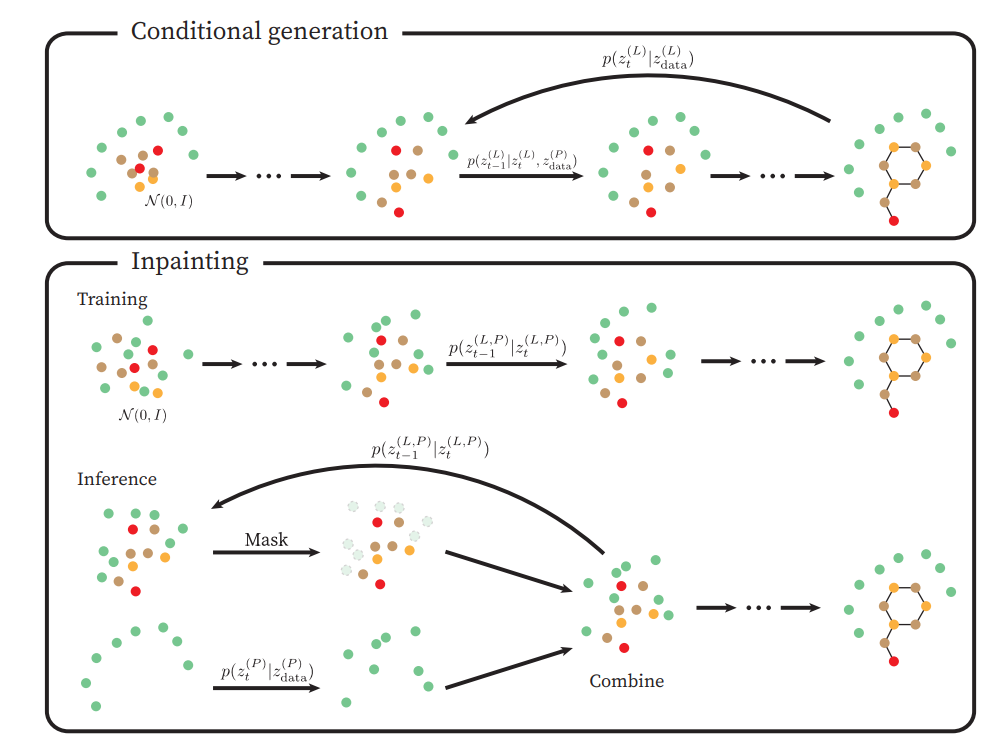
在条件分子的生成设置中,作者在去噪过程的每个步骤中都提供固定的三维上下文。为此,作者用上标 P 表示的蛋白质口袋节点来补充,用上标 L 表示的配体节点点云,并且在整个反向扩散过程中保持不变(图 2)。
作者使用 EGNN 对噪声预测器 进行参数化。为了使用单个 GNN 处理配体和口袋节点,原子类型和残基类型首先通过单独的可学习 MLP 嵌入到一个联合节点的嵌入空间中,并且将坐标更新步骤确定为以下内容:
以确保三维蛋白质上下文在整个 EGNN 层中保持固定。
等变性 在具有3D条件的概率设置中,作者希望在以下意义上确保E(3)-等变:
对正交组 O(3)(包括旋转和反射)的等变性是可以实现的,因为作者是用各向同性的高斯来模拟先验和过渡概率,其中平均矢量在上下文的旋转中进行等价变换。然而,确保平移等变并不容易,因为过渡概率本质上不是平移等变的。为了规避这个问题,作者遵循以前的工作,将整个采样过程限制在系统的质心(CoM)为零的线性子空间。在实践中,这是通过在进行似然计算或去噪步骤之前减去系统的质心来实现的。
joint distribution with inpainting
作为上述条件方法的扩展,作者还提出了一种配体修复方法。修复最初作为一种用于完成图像被遮蔽部分的技术引入,目前已被应用于多个领域,包括生物分子结构。在这里,作者将这个想法扩展到三维点云数据。
作者首先训练一个无条件的 DDPM 来近似配体和口袋节点的联合分布。这使能够在没有额外上下文的情况下对新对进行采样。为了以目标蛋白质袋为条件,需要通过修改概率转换步骤将上下文注入采样过程。在扩散步骤t-1, 蛋白质口袋和配体相组合的潜在表示,是从口袋的前向噪声版本组装而成的,该版本是与 DDPM 根据步骤t的上一个潜在表示预测的配体节点相结合。
以这种方式,作者以相反的顺序从t=T 到t=0遍历马尔可夫链,在每一步中用它们的前向噪声对应物替换预测的口袋节点。等式 (12)限制给定蛋白质口袋的生成过程。由于噪声计划在t=0时将噪声过程的方差降低到几乎为零,因此可以保证最终样本包含蛋白质口袋的不受干扰的表示。
由于该模型被训练以近似配体-口袋对的无条件联合分布,除了将蛋白质和配体节点特征嵌入公共空间的全连接神经网络外,该训练过程与 Hoogeboom 等人开发的无条件分子生成过程相同。已知蛋白质口袋的调节完全委托给采样算法,这意味着这种方法不仅限于配体修复,而且原则上允许在不重新训练的情况下掩盖和替换配体口袋系统的任意部分。
等变性 与条件生成一样,类似的要求也适用于联合概率模型,作者希望有 E(3)不变性,这可以通过等价流从不变先验中获得。与之前的方法相比,主要的复杂问题是缺少参考框架,无法定义有效的平移不变的先验噪声分布 ,因为这样的分布不能整合为一个。因此,有必要将概率模型限制在一个无 CoM 的子空间,如以前的工作中所述。虽然反向扩散过程是为无 CoM 系统定义的,但用等式(11)-(13)中描述的已知口袋的新扩散版本取代预测的口袋节点坐标可能导致非零CoM。为了防止这种情况,作者翻译已知口袋的表示,使其质心与预测的表示相吻合:
然后创建新的组合表示
图 2:条件生成和修复方法之间的比较。条件式模型在蛋白质口袋 的固定上下文中学习去噪分子数据。在修复场景中,模型首先学习近似配体和口袋节点的联合分布。对于采样,通过在每个去噪步骤中将配体的潜在表示与口袋的前向扩散表示相结合来提供上下文。
实验
CrossDocked
作者使用 CrossDocked 数据集并遵循与之前工作相同的过滤和拆分策略。这导致训练集有 100,000个高质量的蛋白质-配体对,测试集有100个蛋白质。使用 MMseqs2通过 30% 的序列同一性完成拆分。
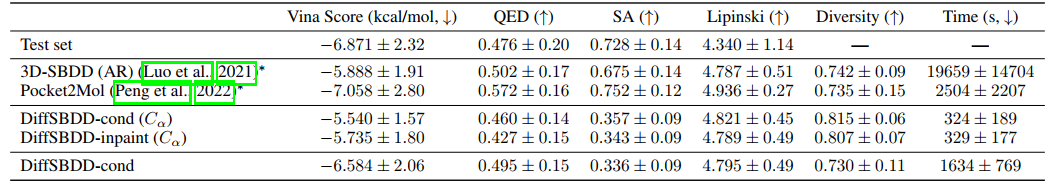
表 1中的实验结果表明,DiffSBDD 可以生成具有预测的高结合亲和力的多种小分子化合物,符合最先进的性能。我们没有看到条件模型和修复方法之间的显著差异。多样性分数可以说是最有趣的,因为这表明与以前的方法相比,作者的模型能够采样更多的化学空间,同时保持高结合性能,这是早期基于结构的先导发现中最重要的要求之一。
表1 对来自CrossDocked测试集的目标生成的分子的评价。*表示重新评估了提供的生成配体
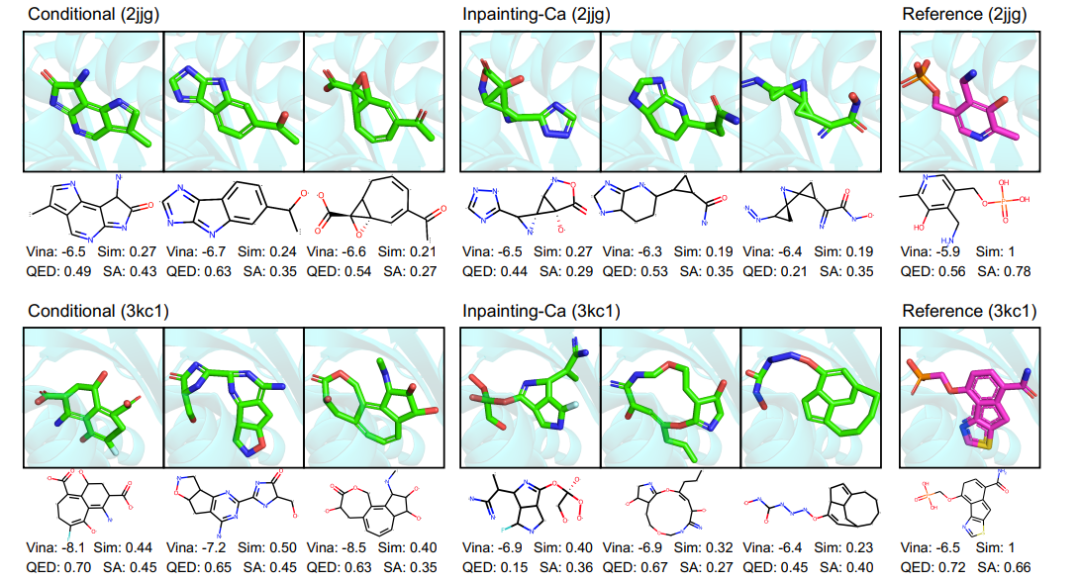
给出了两个靶(2jjg和3kc1)的代表性分子选择,这组分子的策划是为了代表实验中的高得分分子,其中显示了现实的和非现实的motif。值得注意的是,为3kc1生成的第二个分子在与参考配体相同的口袋位置具有相似的三环基序,而参考配体是通过传统的 SBDD 方法设计的,以通过环状系统的形状互补性最大限度地提高疏水相互作用。然而,即使是生成的分子中得分最高的,也存在一些不规则的现象。例如,针对 2jjg(来自Inpainting-)的分子中的大量三角形和 3kc1 的大环将被证明难以合成。
图3 在CrossDocked上训练DiffSBDD模型,并针对氨基转移酶(top,PDB:2jjg)和水解酶(bottom,PDB:3kc1)进行评估。比较了条件和修复方法(分别使用全原子和级蛋白质表示),并展示了每个模型的三个高亲和力分子。’Sim’是生成的配体和参考配体之间的Tanimoto相似度
Binding MOAD
作者评估了在 Binding MOAD中发现的实验确定的蛋白质-配体复合物的方法,这些复合物根据蛋白质的酶委托编号进行过滤和拆分。这会产生 40,354 个蛋白质-配体对用于训练和 130 对用于测试。
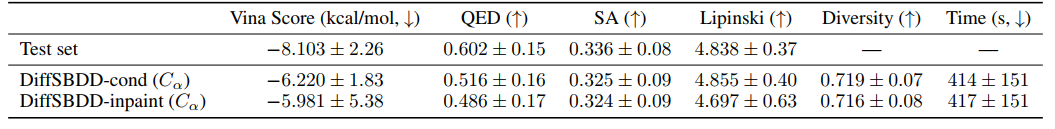
数据集 Binding MOAD与实验确定的结合复合物数据的结果如下表所示。在 130个测试口袋中,每个口袋都产生了 100个有效配体,共产生了 13000个分子。DiffSBDD 生成了高度多样化的分子,但平均对接分数低于该数据集的相应参考配体。
表 2 对来自 Binding MOAD 测试集的目标口袋生成的分子的评估
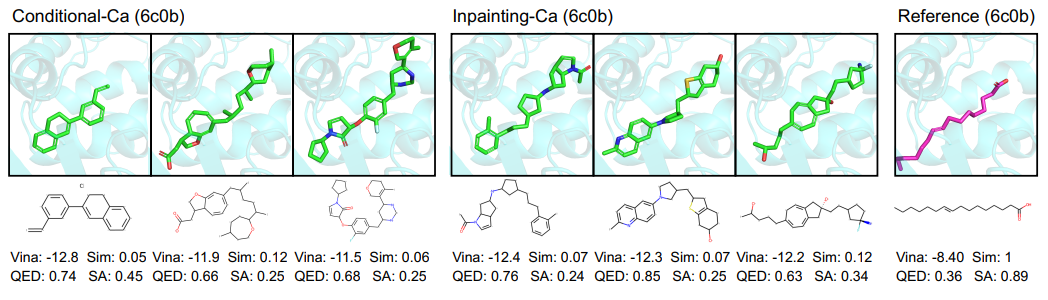
为代表性靶标生成的分子如图 4 所示。靶标 (PDB: 6c0b) 是参与微生物感染和可能的肿瘤抑制的人类受体。参考分子是一种有助于受体结合的长脂肪酸,其可旋转键的数量过多,而氢键供体/受体的数量较少,因此被认为是合适的药物(QED 为 0.36)。然而,提出的模型通过添加由少量可旋转键连接的芳香环来生成类似药物(QED 介于 0.63-0.85 之间)和适当大小的分子,这允许分子采用互补的结合几何形状并且在熵上是有利的(通过减少自由度),这是一种经典的药物化学技术。
图4 在 Binding MOAD 上训练的DiffSBDD模型针对人类受体蛋白(PDB:6c0b)进行了评估。比较了条件和修复方法(两者都是),并给出了每个模型中亲和力最高的三个分子。
结论
在这项工作中,作者提出了 DiffSBDD,一种基于结构的用于药物设计的 E(3)-等变3D 条件扩散模型。作者在合成基准和实验确定的蛋白质-配体复合物的新数据集上证明了 DiffSBDD 在生成新的且多样化的配体方面的有效性,这些配体对给定的蛋白质口袋具有预测的高亲和力。作者证明,在广泛的分子指标上,基于修复的方法可以取得与直接调节相竞争的结果。因此,将这种更通用的策略扩展到全原子口袋表示,有望完成各种其他基于结构的药物设计任务,例如先导优化或接头设计,以及无需重新训练的结合位点设计。
参考资料 Schneuing A, Du Y, Harris C, et al. Structure-based Drug Design with Equivariant Diffusion Models[J]. arXiv preprint arXiv:2210.13695, 2022.