12 月 18 日,在世界人工智能大会发起的 AIWIN 抗新冠人工智能挑战赛的颁奖典礼上,由天士力国际基因网络药物创新中心公司研发的、包含新冠文献智能分析功能的 「星斗云生物医学文献全息智能管理平台」脱颖而出,获得知识图谱类比赛的冠军,并荣获「抗新冠,助科研,AI 赋能者」称号。
![]()
平台链接:http://literature.tasly.com/covid19
在分享现场,天士力基因网络公司的数据总监李旭博士介绍称,天士力的星斗云平台基于多维度生物大数据(海量组学与药物数据及千万级生物医药文献文本等)搭建而成,以网络药理学和人工智能为工具对疾病多维度生物医学知识进行梳理归纳和精准画像,凭借独特设计的 GN-BERT Plus 算法首次提出了知识图谱 + 药物发现的数字孪生平台体系,将海量文献知识的图谱化解析与药物重定位的多维度算法模型交互融合,实现了基于文献的知识图谱在药物发现中的落地应用。
COVID-19 病毒的大流行,激发了全球科学家空前的合作研究,期间产生了大量针对疾病机理、临床诊断、用药治疗层面的科研文献。然而,为了跟踪和了解新的研究内容,研发人员需要在论文的查找、研读和解析上花费大量精力。
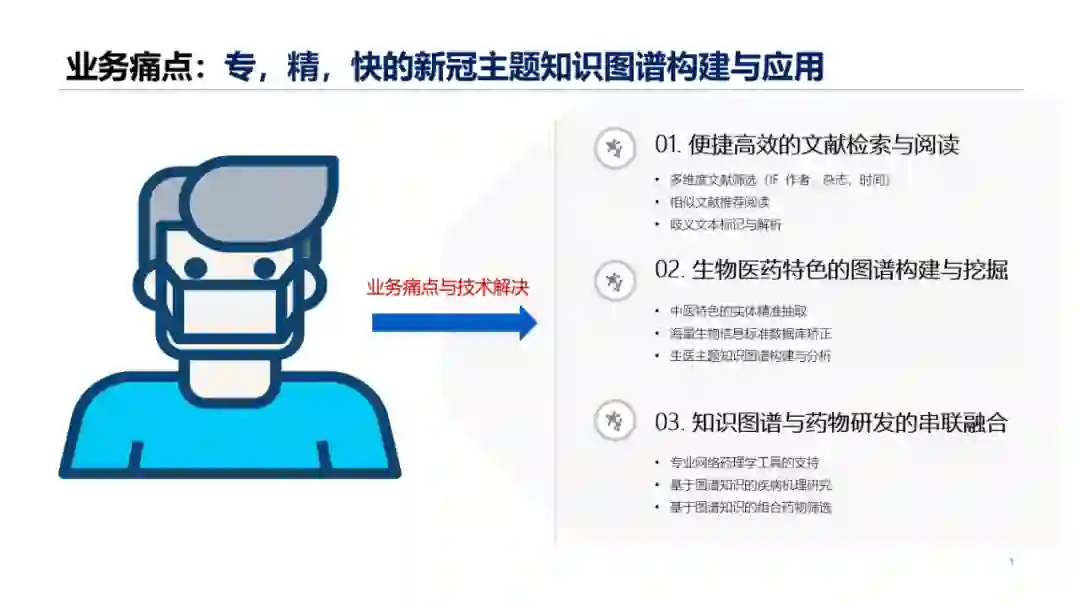
综上,本次参赛的平台基于三方面痛点设计展开。第一,知识图谱使用的痛点,即如何使用知识图谱助力广大医疗、科研和药物开发人员,提高人工搜索和阅读的效率与精度。第二,知识图谱技术的痛点,即如何实现生物医药特色的图谱构建与挖掘,例如中医药特色的实体精准抽取(如化药、中药、症候、方剂、植物等)及海量生物信息标准数据库矫正与图谱化呈现。第三,知识图谱落地的痛点,即如何实现知识图谱与药物研发的串联融合,包括疾病潜在靶点的发现与化合物筛选等。
![]()
下面以知识图谱、文献搜索和网络药理三个部分来介绍冠军组的解决方案和应用。
![]()
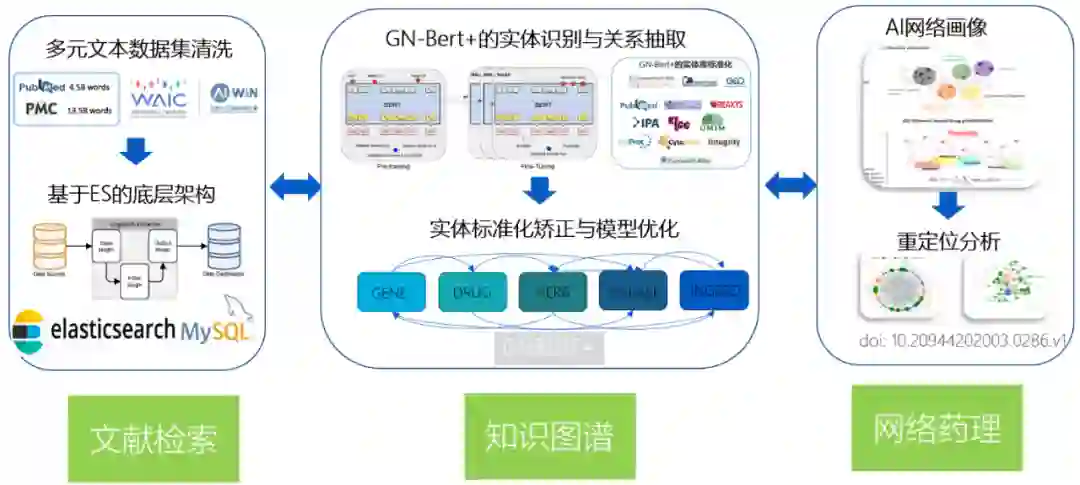
Figure 1:Covid-19 文献智能分析平台的总体框架
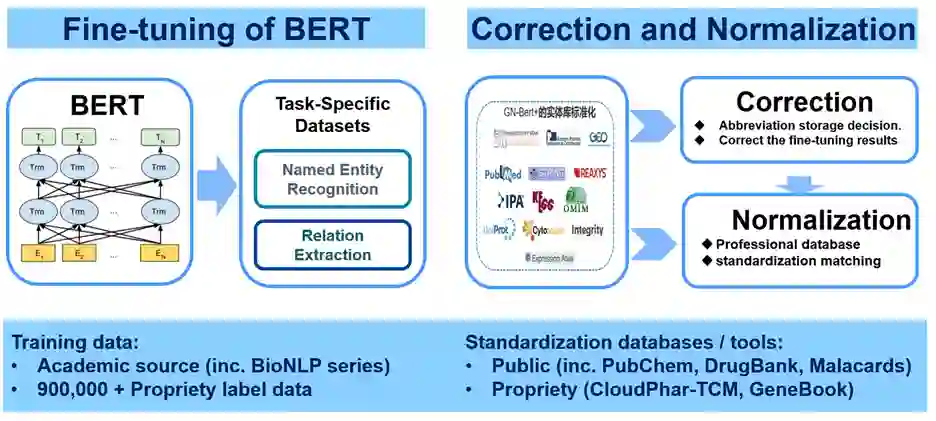
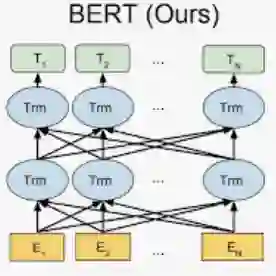
在文本挖掘方面,开发团队用生医领域金标准数据集和自研数据集对 BERT 进行了微调,然后以此为基础,结合星斗云医学特色实体数据库,利用模型和正则表达式匹配补充算法模式,将其整体封装在公司开发的
GN-BERT Plus 引擎
中。
GN-BERT Plus 的实体识别
结合了 4 个策略:(1)借鉴近两年在 BioNLP 科研界最新的工具——BioBERT[2]与 Cross-type[3]做基于机器学习的实体识别;(2)基于正则表达式匹配的方法;(3)缩写存储判定(从文章第一次出现的缩写或缩写参考表里提取缩写对应关系,然后有针对性地判断该文章的缩写);(4)专业数据库标准化匹配。其中,公共的实体标记训练集来自于近十几年来多个科学团队或比赛积累的多个语料集,可从以下网址统一参考或下载:https://github.com/BaderLab/Biomedical-Corpora
该数据库全部是由各领域专家手动标记。同时,团队还拥有自开发的标记数据,特别是针对中文的、中医药相关的,以及一些补充数据。然后,团队从基础 BERT 预训练模型(基于英文维基百科和 BooksCorpus)开始,针对实体识别任务进行 fine-tuning。
GN-BERT Plus 的实体标准化与链接
:团队在生物信息端已经开发了针对人类基因进行去歧义和标准化的 GeneBook 工具(2019SR0589834)及相应数据库、针对中药相关的植物与成分等信息的 CloudPhar 云平台中药数据库(http://cloud.tasly.com/#/tcm/home,目前世界信息最全的中药数据库,投稿中)、针对化学分子的去歧义和标准化工具(主要基于 PubChem 公共数据库)。同时,团队也使用一些公共或权威的数据库,例如 MalaCards 的疾病数据库、DrugBank 的药物数据库等。
GN-BERT Plus 的关系或关联提取
:基于 BERT 进行 fine-tuning 为主、正则表达式匹配为辅来进行关系 / 关联提取。这里与 BioBERT 的算法开发类似,使用了 GAD 和 EU-ADR 的基因 - 疾病关联语料集和 CHEMPROT 的化学分子 - 靶向 - 蛋白 / 基因的语料集;同时使用了内部自己标记的语料集。其余的关系基于词共现(co-occurence)提取。
![]()
Figure 2:GN-BERT Plus 设计原理与支持数据
数据:文献数据分为两部分,一是 AIWIN 比赛共约 30 万篇新冠相关文献;二是来自 GeneNet 公司自有文献库的文献,共有约 3600 万篇中英文开源文献摘要信息,全量文本信息存储在本地搭建的 ElasticSearch 搜索引擎中。此外,天士力 GeneNet 公司自行构建了实体数据库,包含 6 大实体类型,一共 26 万个实体(包括中药方剂 52612 个,西药 8241 个,中药植物 15836 个,中药成分 42845 个,疾病 20388 个,基因 / 蛋白 121329 个)。
架构存储:采用 ElasticSearch (ES)与 MySQL 整合。
知识图谱:利用深度学习方法的 GN-BERT Plus 引擎,内部综合评估结果显示实体识别准确率可达到 93.3%。
图谱交互及可视化:基于 Neo4j+Echarts.js+Vis.js 实现图谱的网络可视化与原文 - 图谱交互,通过对两个前端可视化组件进行封装,结合平台具体功能模块的展示、交互等进行标准化组件输出。
相似论文推荐:针对已选文献给出相似性高的研究论文推荐(Top10 正相关结果),该模块是通过对已选文献与全量文献的词嵌入向量进行遍历相关性分析计算得到,可以较好地满足用户对同一领域知识的快速获取。
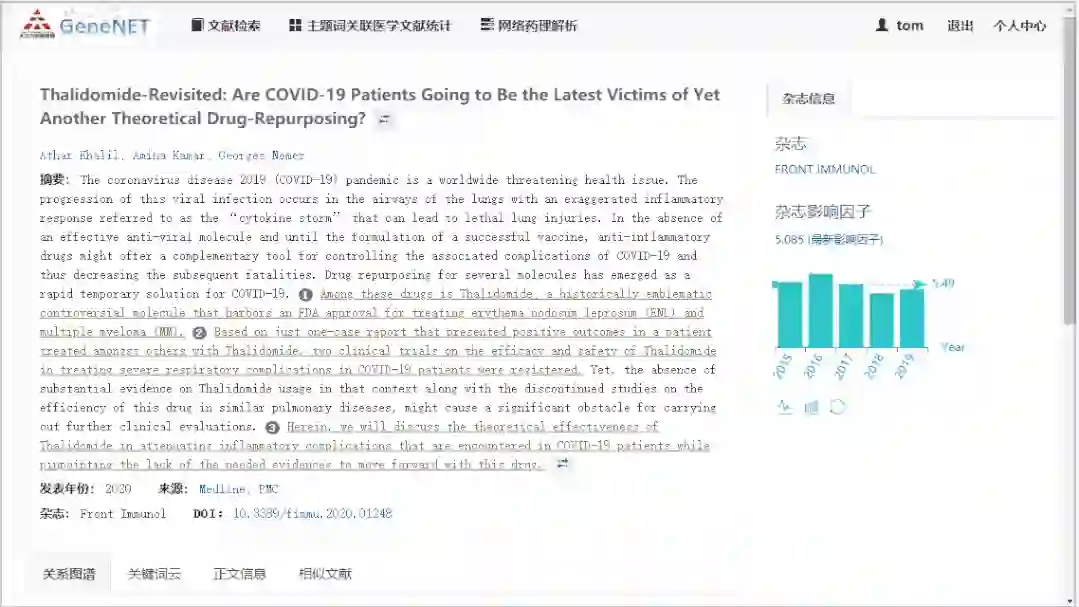
在主要界面,用户可以输入关键词、题目、作者等信息,并通过杂志来源、杂志影响因子和发表年份来做筛选,同时还可以根据影响因子、相关性和发表年份排序。
![]()
点击进入某篇文献后,会有知识图谱显示,并且与文本标记可交互访问,同时还有词云显示、相似文章推荐等功能。
![]()
Figure 4:文章访问页面,知识图谱与文本标记交互
而且,平台提供经过 GN-BERT Plus 挖掘后的文献,实体和关系的总体统计情况。下图是疾病 SARS-Cov-2(也就是 Covid-19,GN-BERT Plus 系统会认为其在 “疾病” 实体上为同义词)关联基因的统计信息。毫无意外,提到 ACE2 与 SARS-Cov-2 之间关系的文献数量最多。通过点击链接,即可得到支持两者关系的所有文献。
![]()
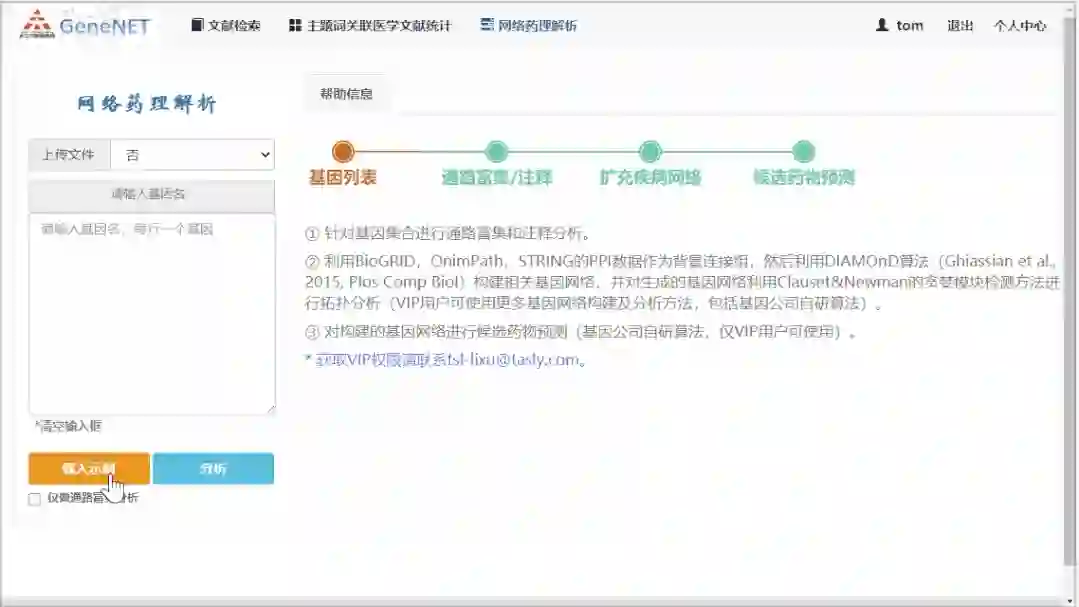
基因列表输入:网络药理学通常会从一个基因列表出发。例如,在 Covid-19 的研究场景里,可以使用上图显示的有足量文献支持的相关基因列表。
通路富集分析:这里使用生物信息常用的信号通路富集方法,对 KEGG 数据库的信号通路集合进行分析。具体使用的是 ClusterProfiler(https://bioconductor.org/packages/release/bioc/html/clusterProfiler.html)工具。
疾病网络构建:利用 BioGRID,OnimPath,STRING 的 PPI 数据作为背景连接组,然后利用 DIAMOnD 算法 [4] 构建相关基因网络,使网络包含的基因数 3 倍于输入基因数,并对生成的基因网络利用 Clauset&Newman 的贪婪模块检测方法进行拓扑分析。
![]()
[1]S. Nabirotchkin, A.E. Peluffo, P. Rinaudo, J. Yu, R. Hajj, D. Cohen, Next-generation drug repurposing using human genetics and network biology, Curr. Opin. Pharmacol. (2020) 1–15. https://doi.org/10.1016/j.coph.2019.12.004.
[2]J. Lee, W. Yoon, S. Kim, D. Kim, S. Kim, C.H. So, J. Kang, BioBERT: A pre-trained biomedical language representation model for biomedical text mining, Bioinformatics. 36 (2020) 1234–1240. https://doi.org/10.1093/bioinformatics/btz682.
[3]X. Wang, Y. Zhang, X. Ren, Y. Zhang, M. Zitnik, J. Shang, C. Langlotz, J. Han, Cross-type biomedical named entity recognition with deep multi-task learning, Bioinformatics. (2019). https://doi.org/10.1093/bioinformatics/bty869.
[4]S.D. Ghiassian, J. Menche, A.L. Barabási, A DIseAse MOdule Detection (DIAMOnD) Algorithm Derived from a Systematic Analysis of Connectivity Patterns of Disease Proteins in the Human Interactome, PLoS Comput. Biol. 11 (2015) 1–21. https://doi.org/10.1371/journal.pcbi.1004120.
© THE END
转载请联系本公众号获得授权
投稿或寻求报道:content@jiqizhixin.com