近期,预训练的基础模型在多个领域都实现了显著的进步。但在分子机器学习中,由于数据集经常是手工策划的,通常比较小,因此缺乏带标签特征的数据集和管理这些数据集的代码库,这都限制了基础模型的发展。在这项工作中,我们介绍了七个按大小划分为三个明确类别的新数据集:ToyMix、LargeMix 和 UltraLarge。这些数据集在监督标签的规模和多样性上都为分子学习设定了新的标准。它们涵盖了近1亿分子和超过3000个稀疏定义的任务,总计超过130亿个量子和生物性质的独立标签。相比之下,我们的数据集包含的数据点是广泛使用的 OGB-LSC PCQM4Mv2 数据集的300倍,和仅限于量子的 QM1B 数据集的13倍。此外,为了支持基于我们提出的数据集的基础模型的发展,我们展示了Graphium图机器学习库,它简化了为多任务和多层次分子数据集构建和训练分子机器学习模型的过程。最后,我们提供了一系列基线结果,作为在这些数据集上进行多任务和多层次训练的起点。从经验上看,我们观察到,在资源有限的生物数据集上的性能通过在大量的量子数据上进行训练也得到了提高。这表明在多任务和多层次训练的基础模型上,并对资源受限的下游任务进行微调,可能有潜力。
https://www.zhuanzhi.ai/paper/8bd15b3e2a1a4aa7b8d48bdab77042c3
图形和几何深度学习模型已经成为近年来机器学习在药物发现中成功的关键组成部分(Gasteiger et al.; Masters et al., 2023; Rampášek et al., 2022; Ying et al., 2021)。这些方法在分子表示学习(2D (Rampášek et al., 2022)、3D (Gasteiger et al.; 2021) 和 4D (Wu et al., 2022) 的情况下)、活性和性质预测 (Huang et al., 2021)、力场开发 (Batatia et al., 2022)、分子生成 (Bilodeau et al., 2022) 以及原子相互作用的建模 (Corso et al., 2022) 中展现了相当的性能。和其他深度学习方法一样,它们需要大量的训练数据来实现高模型精度。然而,在当前的治疗文献中,大多数训练数据集的样本都是有限的 (Huang et al., 2021)。令人心动的是,自监督学习和自然语言处理 (NLP) (Brown et al., 2020a; Liu et al., 2023b) 和计算机视觉 (CV) (Dosovitskiy et al.) 中的基础模型的最新进展已经极大地提高了深度学习的数据效率。事实上,通过预先投资于使用大量数据预训练大模型,一次性的成本,事实证明,学到的归纳偏见降低了下游任务的数据需求。
继这些成功之后,许多研究已经探讨了大型分子图神经网络的预训练及其在低数据分子建模中的益处 (Lin et al., 2022; Méndez-Lucio et al., 2022; Zhou et al., 2023)。然而,由于大型带标签的分子数据集的稀缺,这些研究只能利用自监督技术,如对比学习、自动编码器或去噪任务 (Hu et al., 2020a; Xia et al., 2023)。通过从这些模型中微调的低数据建模努力,迄今为止,只实现了自监督模型在NLP和CV中所取得的部分进展 (Sun et al., 2022)。这部分是由于分子和它们的构象体作为图形的未确定性解释的,因为它们的行为是环境依赖的,并主要由量子物理规定。例如,众所周知,结构相似的分子可能具有完全不同的生物活性,这被称为活性断层,这限制了仅基于结构信息的图形建模 (van Tilborg et al., 2022)。我们认为,为分子建模构建有效的基础模型需要使用量子力学 (QM) 描述和生物环境依赖数据的监督训练。
**贡献 **
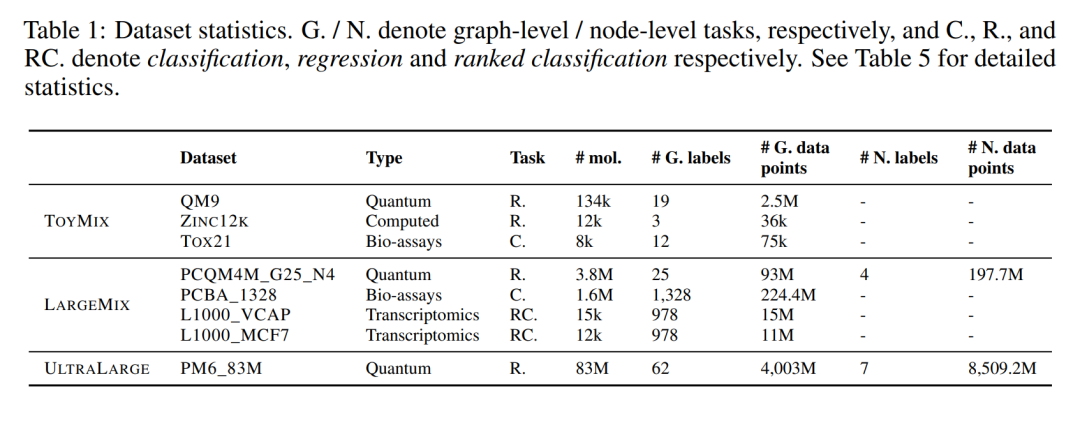
本研究以三种方式推进了分子研究。首先,我们引入了一个新的多任务数据集家族,这些数据集的规模比当前的技术水平要大得多。其次,我们描述了一个名为Graphium的图机器学习库,以便在这些大型数据集上进行高效训练。第三,我们实现了一系列基线模型,支持在多种任务集合上进行训练的价值。 数据集我们引入了三个大型且精心策划的多标签数据集,这些数据集覆盖了近1亿个分子和超过3000个稀疏定义的任务,总计超过130亿个单独的标签,目前是其种类中最大的。这些数据集是为基础模型的监督训练而设计的,通过结合表示量子和生物属性的标签,这些属性是通过模拟和湿实验室实验获得的。这些标签也是多层次的,包括节点级和图级任务。标签的多样性促进了高效的迁移学习,并使得在广泛的下游分子建模任务中提高基础模型的泛化能力成为可能。为了创建这些全面的数据集,我们仔细策划并增强了现有数据,加入了额外的信息。因此,我们的收藏中的每一个分子都伴随着其量子力学 (QM) 属性和/或生物活动的描述。QM属性涵盖能量、电子和几何方面,使用各种先进的方法计算,包括像B3LYP (Nakata & Shimazaki, 2017) 这样的密度泛函理论 (DFT) 方法,以及像PM6 (Nakata et al., 2020) 这样的半经验方法。在生物活性方面,我们的数据集包括从剂量反应生物测定、基因表达谱分析和毒理学谱分析获得的分子签名,如图1所示。联合建模量子和生物效应促进了描述分子的复杂环境依赖属性的能力,这些属性从通常有限的实验数据集中提取是不可行的。
**Graphium库 **我们开发了一个名为Graphium的综合图机器学习库,以便在这些广泛的多任务数据集上进行高效训练。这个新颖的库简化了建立和训练分子图基础模型的过程,通过结合特征集合和复杂的特征相互作用。通过将特征(位置和结构)和表示视为基本的构建模块,并实现最先进的GNN层,Graphium克服了现有框架带来的挑战,这些框架主要是为具有有限节点、边和图特征之间相互作用的顺序样本而设计的。此外,通过提供诸如数据集组合、缺失数据处理和联合训练等功能,Graphium处理了在大型数据集合上训练模型的关键且通常复杂的工程,以简单且高度可定制的方式。
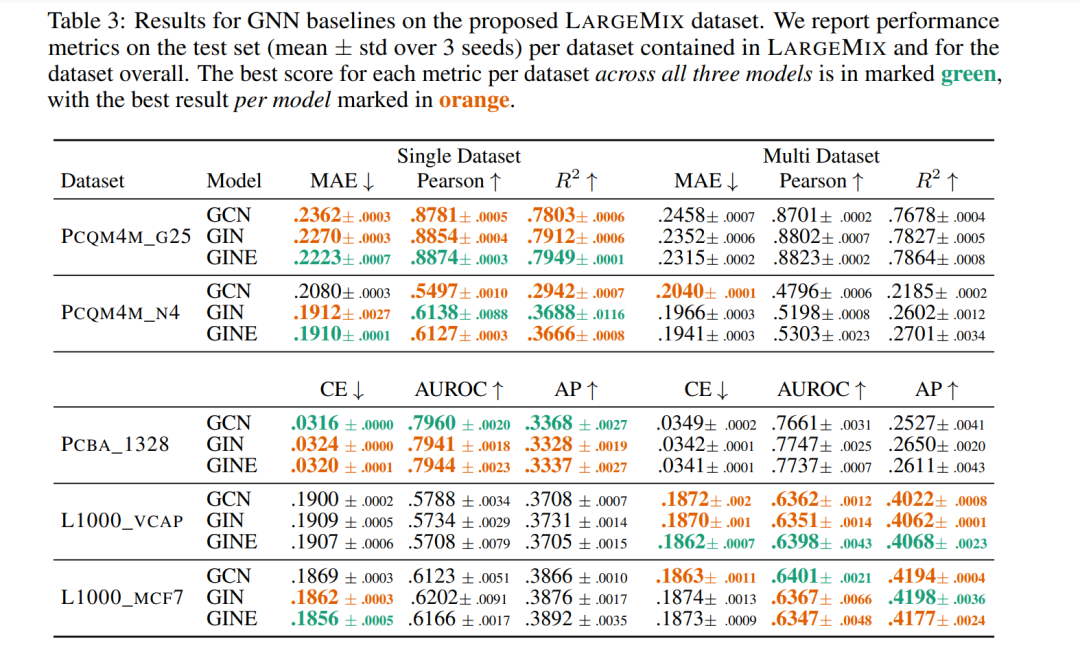
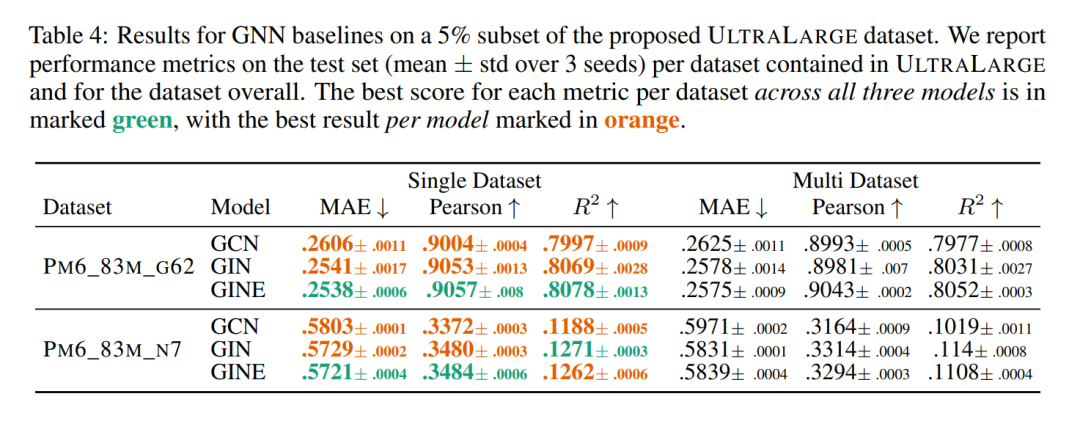
**基线结果 **我们在单数据集和多数据集情境中训练了一系列模型,用于所提出的数据集混合。这些结果提供了坚实的基线,可以帮助指导这些数据集的未来用户,但也给出了在这种多数据集范式中训练的价值的一些指示。具体来说,这些模型的结果显示,与大型数据集结合训练可以显著提高低资源任务的训练效果。总之,我们的研究介绍了迄今为止最大的2D分子数据集,专门设计用于训练可以有效理解分子的量子属性和生物适应性的基础模型,因此可以微调到广泛的下游任务。此外,我们开发了Graphium库,简化了这些模型的训练过程,并展示了一系列基线结果,突显了所提出的数据集和库的有效性。
构建数据集
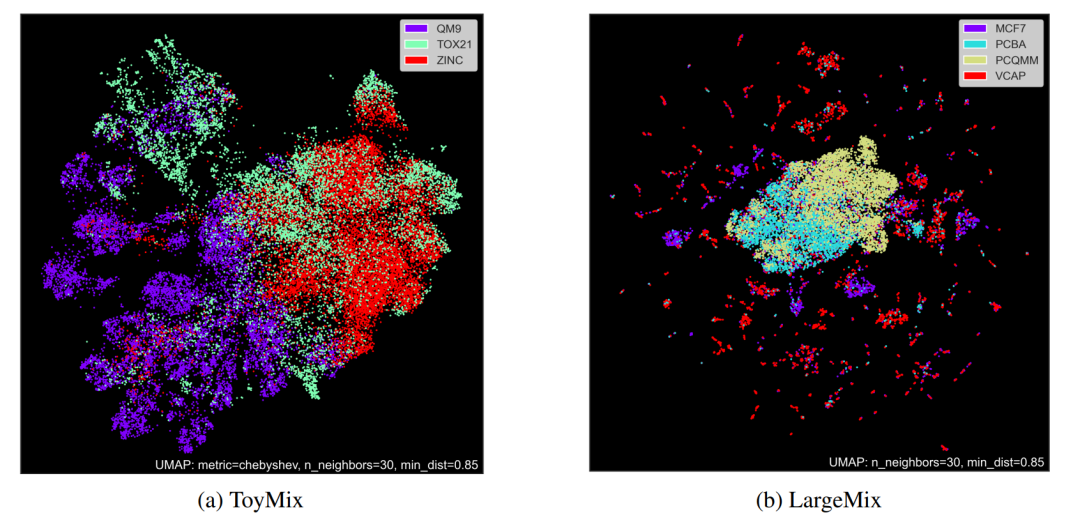
而不是提出一个单一任务数据集,我们提出一个数据集的集合或“混合”,使得在与量子化学和生物化学有关的任务中实现多任务学习,并建立基础模型。数据集在图1中进行了可视化,并在表1中补充了统计数据。有关数据集许可证和可用性的详细信息,请参见附录B.2和可复制性部分,有关数据集的深入了解,请参见附录D。 首先,我们提出一个较小的TOYMIX数据集,以便快速迭代架构,同时也提供有价值的见解。其次,我们提议LARGEMIX,一个精心策划的数据集混合,包含数百万化学化合物的数据,有数千个生物或量子标签。最后,我们提出超大的PM6_83M数据集,它以超过120亿个为8300万分子定义的标签,推动了数据集规模的极限。在以下部分,将详细描述这三个数据集类别。
Graphium库
药物发现是一个拥有丰富、多面的数据集的领域。数据通常是天然的多任务,包含在各种级别的标签信息,从节点和边缘到节点对和整个图形。因此,在这样复杂的数据集上训练模型需要一种共同学习多个任务级别的策略,这是一个独特的挑战。为了利用这些数据集混合中所包含的丰富信息,并从它们的规模中受益,需要专门的软件来促进以多任务方式进行有效的培训。为此,我们引入了Graphium库,它是专门为分子领域内的大规模多任务和多级机器学习模型而设计的。
Graphium库的关键特性在此总结,详细信息可在附录E中找到,文本中指出了相关部分。整个培训程序在模块化配置文件中定义,例如这允许用户交换架构、数据集或指标以进行快速实验。Graphium库的核心新特性是多级多任务学习,它便于通过模块化的数据加载、任务头和损失函数,在多个具有不同和稀疏标签的数据集上训练模型。更多细节可以在附录E.1中找到。灵活和模块化的建模在附录E.2中详细说明。位置编码方法,为子图内的位置提供节点级别的信息,对于许多先进的分子模型是不可分割的。Graphium中提供了关键方法,包括随机漫步和拉普拉斯特征向量,这在附录E.3中详细说明。标签标准化(附录E.4)、排名分类损失(附录E.5)和缺失数据的处理(附录E.6)是该库的重要细节,有助于结合多个数据来源在一系列任务上,以及数据集中不可避免的稀疏性。为了使大型模型的培训资源高效,很多注意力都集中在对小型模型调整模型参数并将这些应用到大型模型上。其中一个名为µP的方法在Yang等人(2022)中引入,包括在Graphium中,以减少超参数调优的成本。附录E.7中提供了更多细节。该库支持CPU、GPU和IPU [§E.8]硬件以加速训练。进一步的库优化在附录E.9中详细说明。
基线模型的实验
为了在具有数千个标签的多任务设置中展示Graphium库的能力,我们使用3种流行的GNNs,即GCN (Kipf & Welling, 2017)、GIN (Xu等,2019) 和 GINE (Hu等,2020a),使用简单的超参数扫描运行了一套标准基线。对每个模型进行了基本的超参数扫描,并使用多个随机种子进行初始化,为将来的实验提供了一个性能基线进行评估。
除了与数据集一起提供的节点特征(和GINE的边缘特征)外,我们还使用图的Laplacian的前8个特征值和特征向量作为节点级PE。结合特征值和特征向量,并使用MLP进行嵌入,提供了一个由图的频谱属性所决定的PE (Kreuzer等,2021)。我们进一步使用随机行走返回概率(即,随机行走在特定节点开始并在k∈N步后返回到所述节点的概率),再次,与MLP编码器结合使用 (Dwivedi等,2021)。这可以指示分子内部的循环的存在,这在某些分子任务的背景下可能是有信息的,例如,在ZINC数据集上对溶解度(logP)得分进行回归 (Dwivedi等,2020)。
结论
在这项工作中,我们提出了一个前所未有的分子数据集合,为有监督的学习提供了大量的数据点,从而显著增强了药物发现领域研究的可用资源。这些数据集自然地具有多任务和多级的特点,为机器学习模型带来了独特的挑战。为了促进对这些复杂数据集的研究,我们推出了Graphium库,一个专为处理和高效加载大规模分子数据并在各种任务级别上进行多任务学习的框架。该库利用我们的数据集所呈现的独特特点和属性,为分子学习的大型基础模型的训练提供了支持。 此外,我们提供了一些关于我们提出的数据集的基线结果,并显示小型生物数据集在接受大量量子数据的训练时的性能会得到提高。我们假设这种改进的泛化将帮助基础模型在微调到药物发现中常遇到的低资源下游任务时表现良好。