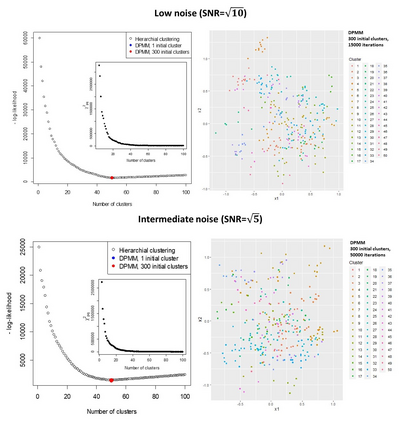
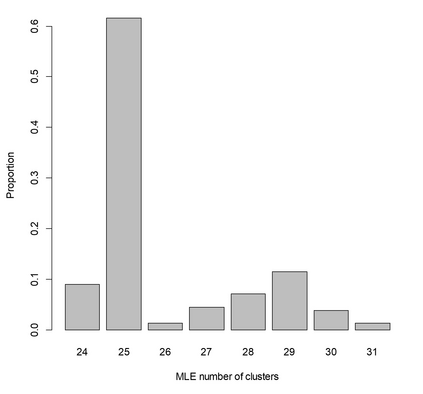
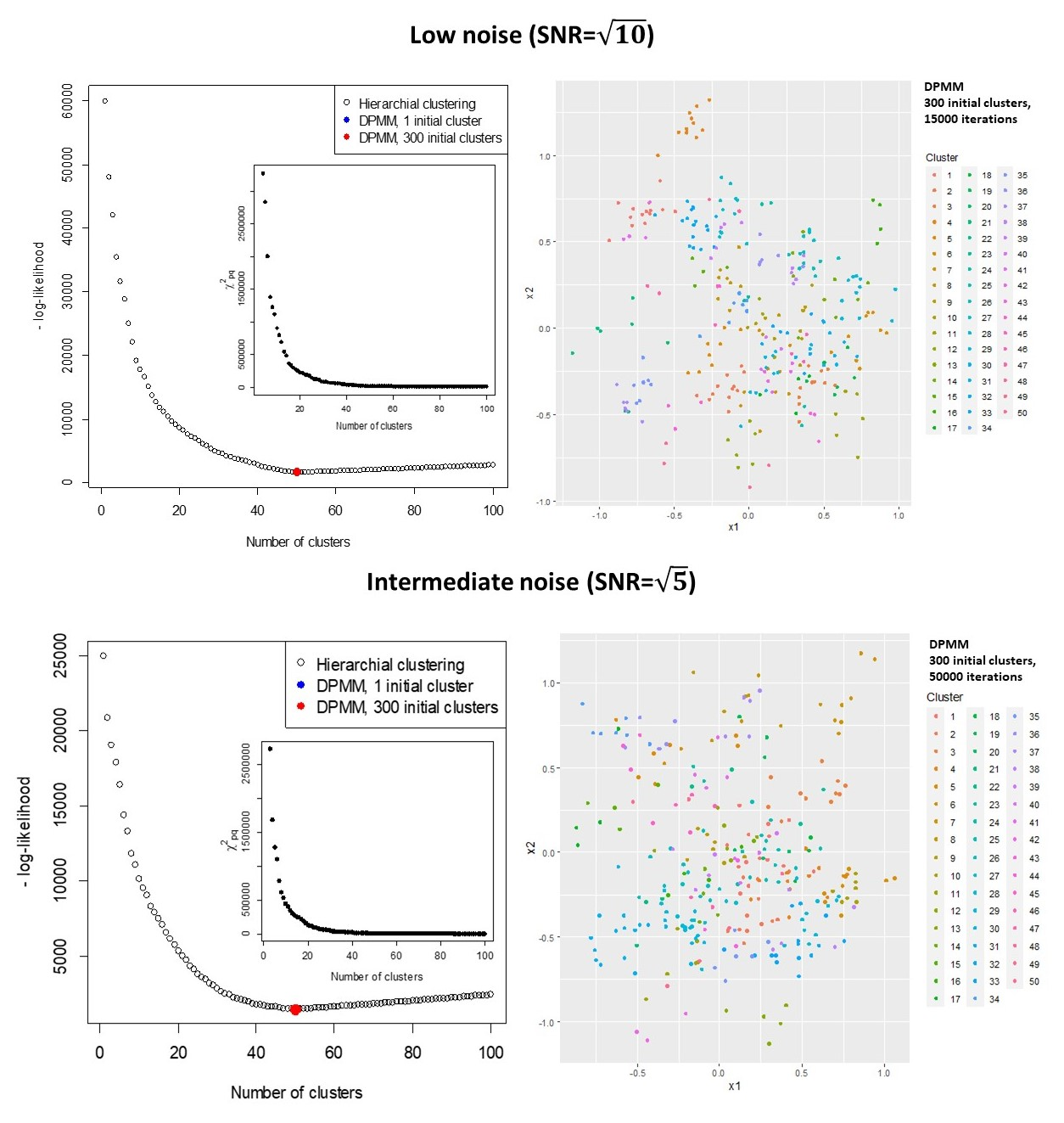
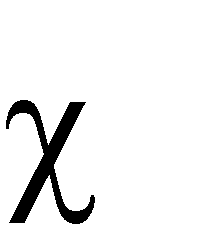A recent UK Biobank study clustered 156 parameterised models associating risk factors with common diseases, to identify shared causes of disease. Parametric models are often more familiar and interpretable than clustered data, can build-in prior knowledge, adjust for known confounders, and use marginalisation to emphasise parameters of interest. Estimates include a Maximum Likelihood Estimate (MLE) that is (approximately) normally distributed, and its covariance. Clustering models rarely consider the covariances of data points, that are usually unavailable. Here a clustering model is formulated that accounts for covariances of the data, and assumes that all MLEs in a cluster are the same. The log-likelihood is exactly calculated in terms of the fitted parameters, with the unknown cluster means removed by marginalisation. The procedure is equivalent to calculating the Bayesian Information Criterion (BIC) without approximation, and can be used to assess the optimum number of clusters for a given clustering algorithm. The log-likelihood has terms to penalise poor fits and model complexity, and can be maximised to determine the number and composition of clusters. Results can be similar to using the ad-hoc "elbow criterion", but are less subjective. The model is also formulated as a Dirichlet process mixture model (DPMM). The overall approach is equivalent to a multi-layer algorithm that characterises features through the normally distributed MLEs of a fitted model, and then clusters the normal distributions. Examples include simulated data, and clustering of diseases in UK Biobank data using estimated associations with risk factors. The results can be applied directly to measured data and their estimated covariances, to the output from clustering models, or the DPMM implementation can be used to cluster fitted models directly.
翻译:最近的一项英国生物银行研究将一个156个参数化模型集中在一起,将风险因素与常见疾病联系起来,以找出共同的病因。参数模型往往比集成数据更熟悉和易解,可以建立先前的知识,对已知的混杂者进行调整,并使用边际化来强调关注参数。估计包括一个(约)通常分布的、且具有共性的最大相似性估计值(MLE) 。组合模型很少考虑通常无法找到的数据点的共变点。在这里,组群模型可以说明数据的共变情况,并假定组群中的所有 MLE 都是一样的。日志相似性精确地按照安装的参数计算,通过边际化来消除未知的群集手段。这个程序相当于计算Bayesian Inforcerication(MLI),并且可以用来评估特定组群集算的最佳组合数。日值术语可以用来惩罚差和模型复杂程度,并且可以最大化地确定组群集中的所有MLE。结果也可以直接使用一个比数性数据模型。结果通常用来使用一个比标准来计算。